{{cell.content}}





撰文 | 十一月
人类大脑在妊娠中期会经历快速的发育,从神经干细胞和祖细胞分化形成神经元、少突胶质细胞和星形胶质细胞等。但是目前还缺乏精确的细胞类型纯化和功能性研究。
近日,美国斯坦福大学Irving L. Weissman研究组与Rahul Sinha研究组合作在Cell发表了文章Purification and characterization of human neural stem and progenitor cells,通过细胞表面标记物发展了对人脑神经干细胞和祖细胞的纯化以及分类方式,为人类神经发育过程中不同细胞类型的功能研究提供了一个新的框架。

人类大脑结构错综复杂,存在着数十亿不同特性的细胞。在发育中的大脑皮层中,神经干细胞和祖细胞分化形成三种主要的神经谱系,神经元、少突胶质细胞和星形胶质细胞。最近单细胞技术为神经转录组多样性提供了高时空分辨率的数据。但是神经干细胞和祖细胞在临床移植等方面的应用价值,最终是由它们的功能进行定义的,特别是自我更新和分化潜能。因此,作者们希望开发一种通过荧光激活细胞分选纯化不同神经干细胞和祖细胞亚群的方法,从而对不同细胞类型进行纯化以及功能性鉴定。
为了实现这一目标,作者们使用高维流式细胞分选以及单细胞转录组对妊娠中期GW17-19的人脑进行神经干细胞和祖细胞类群的分类和功能刻画。作者们利用的是细胞表面标记也就是免疫表型组合表达的方式对单个细胞进行分选,从而达到严格的分选纯度。为了纯化不同的神经干细胞和祖细胞组分,作者们使用机械以及酶解方式将脑组织裂解变成单细胞悬液。随后用一组抗体进行染色,包括CD133、CD24、CD90、CXCR4、EGFR、PDGFRA以及非神经相关的标记物CD45、CD31、CD34、CD105以及CD235a。之后使用流式细胞分选对免疫染色的细胞悬液进行分析。作者们共使用了超过12个该胎龄的样本,在分选后将细胞放入96孔板,再使用Smart-seq2或Smart -seq3进行全长单细胞RNA-seq【1-2】(图1)。
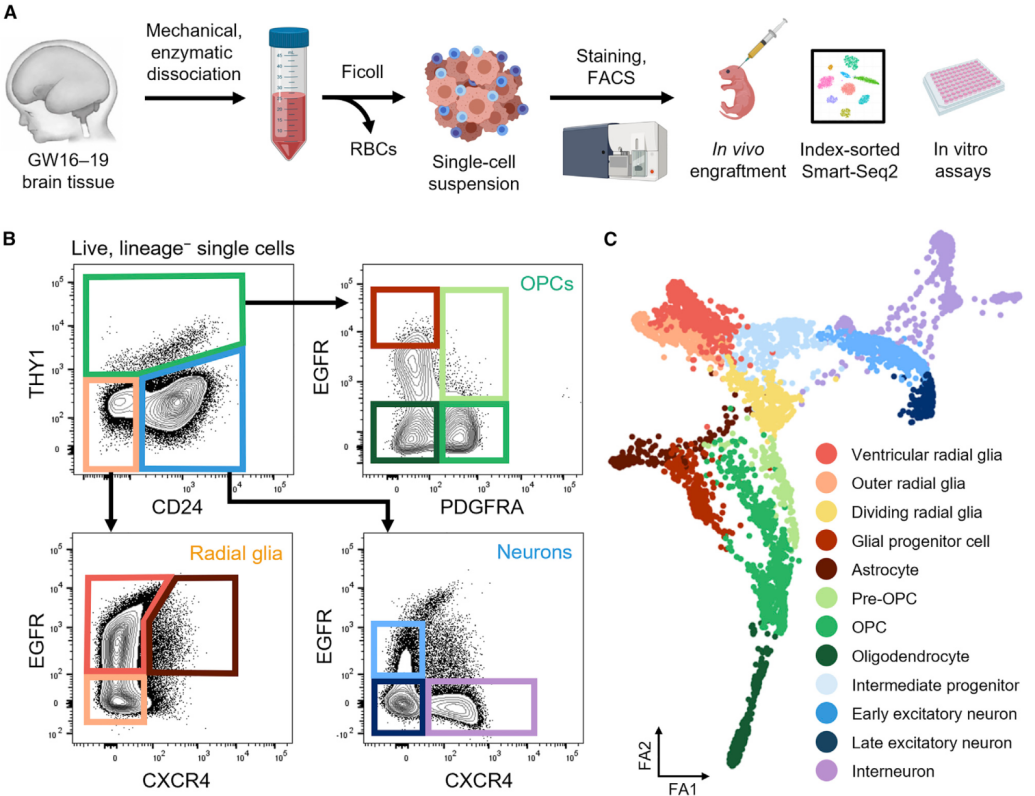
图1 工作流程以及细胞图谱
通过转录组学定义的细胞类型包括vRG、oRG、Acs、pre-OPCs、OPCs、Ols、IPCs以及ExN和InN(图1)。通过转录表达特征与细胞表面标记物进行重叠,作者们发现通过CD24-THY1-/lo门分类,EGFRhi群细胞富集vRG,而EGFP-的细胞群则是oRG。免疫组学也确定了EGFR表达的脑区,因此EGFR能够对vRG以及oRG进行区分。另外,PAX3表达在AC细胞谱系中,说明PAX3在早期神经干细胞与AC谱系分歧之间发挥重要作用。
进一步地,为了对推定得到的神经干细胞和祖细胞亚群进行功能性分析,作者们对不同细胞类型的自我更新和分化能力分别在体内和体外进行检测。CD24-THY1-/lo神经干细胞在体外能够形成神经球(Neurospheres),通过稀释分析实验作者们证实了CD24-THY1-/lo门分选后,EGFRhi群细胞相较于低表达EGFR的细胞具有更高的神经球发育起始能力。为了对推定的神经干细胞在体内的功能进行鉴定,作者们对分离的细胞进行小鼠体内的移植实验。在移植六个月后,作者们对移植体的特征进行分析,发现移植的神经干细胞能够迁移和定植到不同脑区中并形成三种主要的神经元类型。
细胞分选中THY1hi表达的细胞是推定的少突胶质细胞祖细胞OPCs,这些细胞广泛表达OPCs标记物OLIG1、OLIG2、SOX10等。这些细胞在体外会形成少突胶质细胞祖细胞形态,而在体内移植后则只会命运呈递形成少突胶质细胞OLs。另外,通过单细胞数据分析,作者们鉴定出了一种双潜能的胶质前体细胞,既能够分化形成AC也能够形成OL。体外培养和体内的移植实验证明了该群细胞双潜能的存在。
最后作者们对不同脑区中的细胞进行分析,发现细胞表面的标记物在不同区域中是保守的。通过对不同细胞表面标记物的分析,作者们发现了352种额外的表面标记物能够对神经干细胞和祖细胞的身份进行定义,并将该特征数据库称为神经干细胞和祖细胞的表面标记物组(Surfaceome)。
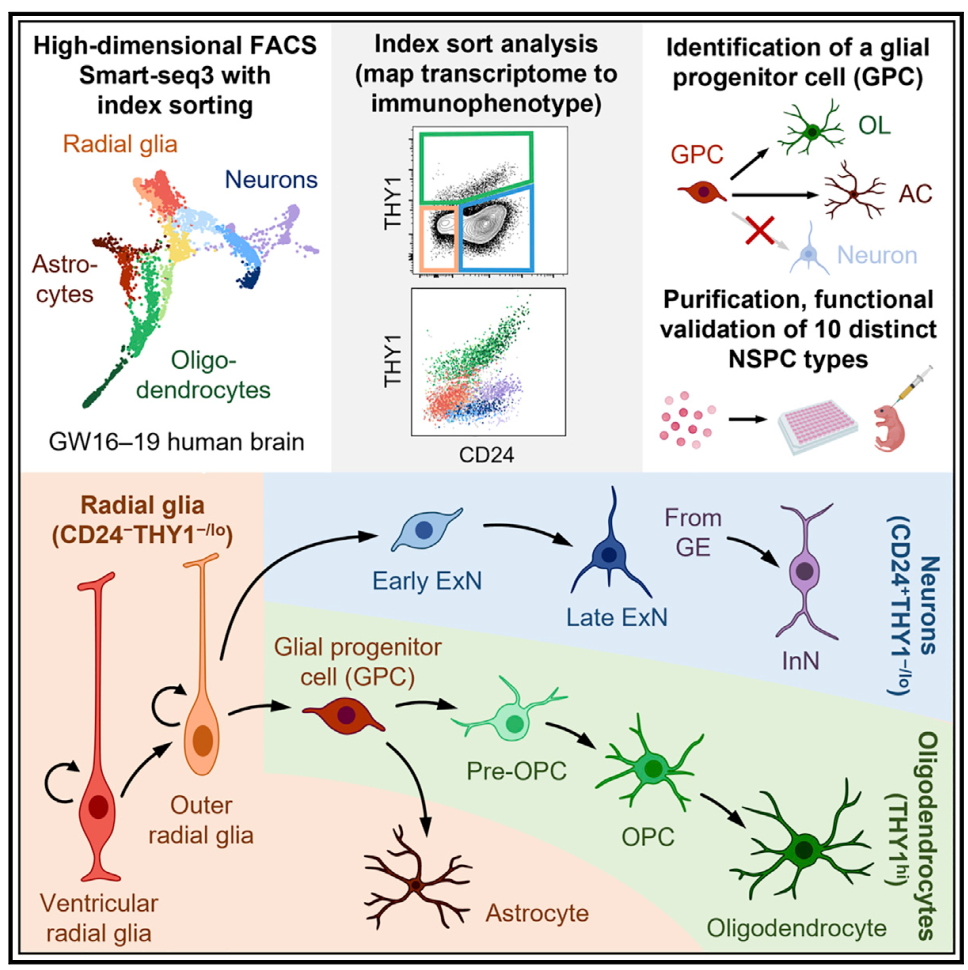
图2 工作模型
总的来说,作者们的工作通过对的发育中人脑细胞进行表面标记的分选和纯化,对不同细胞类型进行了定义。进一步地,作者们通过纯化细胞的体外培养以及小鼠脑内移植确定了不同类型神经干细胞和祖细胞的自我更新与分化潜能。这一工作为人类发育时期大脑细胞类型的功能性定义提供了新的框架。
原文链接:
https://doi.org/10.1016/j.cell.2023.02.017
参考文献
1. Picelli, S., Faridani, O.R., Bjo¨ rklund, A.K., Winberg, G., Sagasser, S., and Sandberg, R. (2014). Full-length RNA-seq from single cells using Smartseq2. Nat. Protoc. 9, 171–181. https://doi.org/10.1038/nprot.2014.006.
2. Hagemann-Jensen, M., Ziegenhain, C., Chen, P., Ramsko¨ ld, D., Hendriks, G.J., Larsson, A.J.M., Faridani, O.R., and Sandberg, R. (2020). Single-cell RNA counting at allele and isoform resolution using Smart-seq3. Nat. Biotechnol. 38, 708–714. https://doi.org/10.1038/s41587 020-0497-0.


想了解更多精彩内容,快来关注BioArt生物艺术
特别声明:本文为人民日报新媒体平台“人民号”作者上传并发布,仅代表作者观点。人民日报提供信息发布平台。

写下你的评论
{{cell.content}}
{{item.content}}
{{item.content}}