{{cell.content}}





撰文 | 我的闺蜜老红帽
儿童实体瘤,尤其是高风险的中枢神经系统肿瘤,目前是小儿肿瘤致死的主要原因之一。尽管学界在小儿肿瘤基因组学分析领域已经取得了长足进展,但对于低生存率小儿肿瘤的精准医疗方面仍旧落后于成人肿瘤治疗。近期的工作指出,小儿脑部肿瘤呈现出与极为特异且临床相关的多种分子亚型【1-3】,这使得相关研究以及转化更为复杂,也使得学界逐渐关注各亚型的特异性遗传驱动因素、活化信号以及相关功能。因此,在多维度构建和研究小儿肿瘤模型势在必行【4】。
大规模的基于细胞系的肿瘤组织,比如Cancer Cell Line Encyclopedia和Human Cancer Models Initiative,是研究成人肿瘤的重要资源【5-7】。相关成果已于近期被应用于小儿肿瘤中,比如初代儿科关系依赖图(dependency map,简称DepMap),平均每种肿瘤最多收集6个实体瘤细胞系,共整合了82个小儿实体瘤细胞系的功能基因组数据【8】。因为小儿肿瘤的分子亚型多种多样,尤其是脑部肿瘤,扩大小儿肿瘤细胞系集合,以及在深度和广度两个维度建立建立肿瘤数据库并总结突变类型是目前亟需所做的工作。
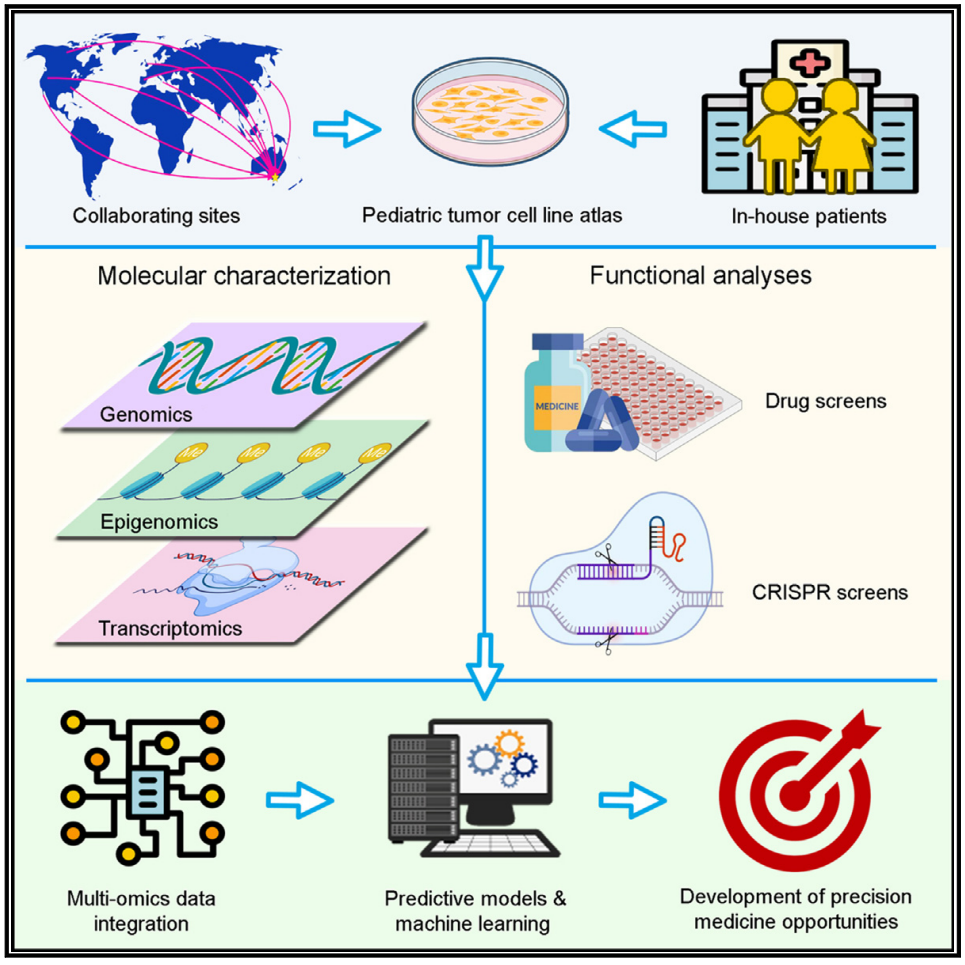
近日,来自澳大利亚Monash University的Ron Firestein研究组在cancer cell上发表题为Generation and multi-dimensional profiling of a childhood cancer cell line atlas defines new therapeutic opportunities的文章,有力推进上述工作,建立了小儿肿瘤模型图谱(Childhood Cancer Model Atlas,简称CCMA)。

作者收集了261个细胞系,包括224个小儿细胞系,分属18种不同颅外(extracranial)和脑部小儿肿瘤类型。作者对其中182个细胞系进行了多组学分析,包括DNA测序、RNA测序和DNA甲基化分析。
与此同时,作者还进行了基于CRISPR-Cas9技术的病理学和遗传学功能分析,并基于上述结果,力图确定小儿特异性诊疗标签以及可行性临床治疗方案。作者所筛选出的多个分子小儿和成人具有明显差异,并且可以作为潜在临床靶点,包括PIK3CA, MCL1, FGFR1。这也说明,关注小儿肿瘤的试验方法可以有效识别临床靶标以及药物靶点。另外,作者还发现多类肿瘤特异性遗传脆弱性(genetic vulnerabilities)位点,比如骨肉瘤(osteosarcoma)的YES1和畸胎瘤样横纹肌样肿瘤(atypical teratoid rhabdoid tumors,简称ATRT)的NSD1。作者还从功能角度深入探讨了多组分类明确、但功能尚不清晰的中枢神经系统肿瘤亚型,包括H3 G34-突变型 弥漫性半球胶质瘤(diffuse hemispheric gliomas,简称DHG)中的TGFBR1/2, H3.3-K27M变化的弥漫性中线胶质瘤(diffuse midline gliomas ,简称DMG)中的HDAC2, 以及I型ATRT中的KMT2A。上述研究思路也可以推广至其它小儿肿瘤类型。
整合多种分子和功能组学分析的方法使得建立特定基因易损性和可能的分子响应之间的关系成为可能。作者构建了小儿肿瘤特异的、包含132个基因和53个可能药物靶点的分子谱。并且,作者进一步确定了SLC5A6可以作为DHFR抑制剂的可行性分子标签。当然,通过小鼠模型和类器官进行进一步验证可以作为下一步的工作重点。目前成人肿瘤所采用的诊疗标签对于小儿肿瘤并不完全适用,而作者的工作在一定程度上填补了这部分空白。
综上所述,作者建立的小儿肿瘤模型图谱主要关注于(1)建立新的小儿肿瘤细胞模型,尤其是高风险、低生存率中枢神经系统肿瘤模型;(2)绘制了上述肿瘤细胞分子特征以及基因易损性图谱。作者在分子水平,阐明了不同小儿肿瘤亚型的特异性分子标签以及信号通路,并且还提出了若干可用于临床的诊疗标签。
原文链接:
https://doi.org/10.1016/j.ccell.2023.03.007
参考文献
1. Mackay, A., Burford, A., Carvalho, D., Izquierdo, E., Fazal-Salom, J., Taylor, K.R., Bjerke, L., Clarke, M., Vinci, M., Nandhabalan, M., et al. (2017). Integrated molecular meta-analysis of 1,000 pediatric high-grade and diffuse intrinsic pontine glioma. Cancer Cell 32, 520–537.e5. https:// doi.org/10.1016/j.ccell.2017.08.017.
2. Torchia, J., Golbourn, B., Feng, S., Ho, K.C., Sin-Chan, P., Vasiljevic, A., Norman, J.D., Guilhamon, P., Garzia, L., Agamez, N.R., et al. (2016). Integrated (epi)-Genomic analyses identify subgroup-specific therapeutic targets in CNS rhabdoid tumors. Cancer Cell 30, 891–908. https://doi.org/ 10.1016/j.ccell.2016.11.003.
3. Cavalli, F.M.G., Remke, M., Rampasek, L., Peacock, J., Shih, D.J.H., Luu, B., Garzia, L., Torchia, J., Nor, C., Morrissy, A.S., et al. (2017). Intertumoral heterogeneity within medulloblastoma subgroups. Cancer Cell 31, 737– 754.e6. https://doi.org/10.1016/j.ccell.2017.05.005.
4. Brabetz, S., Leary, S.E.S., Gro ̈ bner, S.N., Nakamoto, M.W., Sxeker-Cin, H., Girard, E.J., Cole, B., Strand, A.D., Bloom, K.L., Hovestadt, V., et al. (2018). A biobank of patient-derived pediatric brain tumor models. Nat. Med.24, 1752–1761. https://doi.org/10.1038/s41591-018-0207-3.
5. Tsherniak, A., Vazquez, F., Montgomery, P.G., Weir, B.A., Kryukov, G., Cowley, G.S., Gill, S., Harrington, W.F., Pantel, S., Krill-Burger, J.M., et al. (2017). Defining a cancer dependency map. Cell170, 564– 576.e16. https://doi.org/10.1016/j.cell.2017.06.010.
6. Carvalho, D.M., Temelso, S., Mackay, A., Pemberton, H.N., Rogers, R., Kessler, K., Izquierdo, E., Bjerke, L., Salom, J.F., Clarke, M., et al. (2020). Drug screening linked to molecular profiling identifies novel depen- dencies in patient-derived primary cultures of paediatric high grade glioma and DIPG.Cancer Biol. https://doi.org/10.1101/2020.12.29.424674.
7. Corsello, S.M., Nagari, R.T., Spangler, R.D., Rossen, J., Kocak, M., Bryan, J.G., Humeidi, R., Peck, D., Wu, X., Tang, A.A., et al. (2020). Discovering the anti-cancer potential of non-oncology drugs by systematic viability profiling. Nat. Cancer 1, 235–248. https://doi.org/10.1038/s43018-019- 0018-6.
8. Dharia, N.V., Kugener, G., Guenther, L.M., Malone, C.F., Durbin, A.D., Hong, A.L., Howard, T.P., Bandopadhayay, P., Wechsler, C.S., Fung, I., et al. (2021). A first-generation pediatric cancer dependency map. Nat. Genet.53, 529–538. https://doi.org/10.1038/s41588-021-00819-w.


想了解更多精彩内容,快来关注BioArt生物艺术
特别声明:本文为人民日报新媒体平台“人民号”作者上传并发布,仅代表作者观点。人民日报提供信息发布平台。

写下你的评论
{{cell.content}}
{{item.content}}
{{item.content}}